Newsletter DPT Nro. 30
ISSN 2618-236X
Octubre / 2018
Reseña de artículos de integrantes del equipo IUDPT (e.f.)
Target-Pathogen: una herramienta bioinformática para priorizar blancos en patógenos
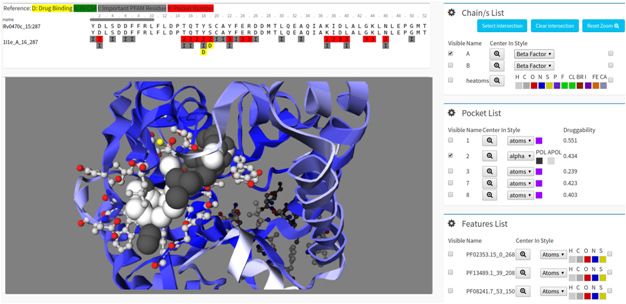
Presentamos seguidamente el resumen de un artículo en cuya autoría participó el Dr. Adrián Turjanski, propuesto como director de la carrera de posgrado Maestría en Bioinformática del proyectado Instituto Universitario DPT (en formación).
El uso de antibióticos viene enfrentando, durante las últimas décadas, el desafío resultante de diversas formas de resistencia desarrolladas por los patógenos microbianos.
Por otra parte, varios proyectos de desarrollo de fármacos han resultado improcedentes por razones que van desde la mala selección de los blancos o dianas (“targets”) hasta la dificultad de diversas firmas farmacéuticas para descubrir nuevos fármacos antimicrobianos. Es ampliamente reconocido que la identificación y validación de blancos apropiados son pasos críticos para el diseño de nuevas drogas.
Los datos genómicos disponibles para los patógenos han creado nuevas oportunidades para el descubrimiento y desarrollo de fármacos para luchar contra esos patógenos, incluyendo nuevas cepas resistentes y multirresistentes. En particular, los datos estructurales deben integrarse tanto con la información genética como con resultados experimentales.
En este sentido, se manifiesta una insuficiencia de recursos “on line” para consolidar los datos genómicos con una amplia base de diversas fuentes, junto con un análisis bioinformático exhaustivo que facilite los procesos de filtrado de datos y ponderación para seleccionar el blanco para el descubrimiento de fármacos.
En el artículo se presenta la base de datos Target-Pathogen (*), diseñada y desarrollada como un recurso en línea que permite integrar y ponderar la información de proteínas, tales como: función, rol metabólico, off-targeting, propiedades estructurales, incluidos la drogodependencia (“druggability”) y la esencialidad. El disponer de tal información facilita significativamente la identificación y priorización de blancos candidatos en patógenos.
Dicha base de datos incluye los genomas de diez (10) microorganismos que afectan –de manera relevante- la salud humana (Mycobacterium tuberculosis, Mycobacterium leprae, Klebsiella pneumoniae, Plasmodium vivax, Toxoplasma gondii, Leishmania major, Wolbachia bancrofti, Trypanosoma brucei, Shigella dysenteriae y Schistosoma Smanosoni) y muestra su aplicabilidad. Pueden cargarse nuevos genomas a requerimiento.
Fuente: “Target-Pathogen: a structural bioinformatic approach to prioritize drug targets in pathogens”. Ezequiel J Sosa; Germán Burguener; Esteban Lanzarotti, Lucas Defelipe; Leandro Radusky; Agustín M Pardo; Marcelo Marti; Adrián G Turjanski; Darío Fernández Do Porto. Nucleic Acids Research, Volume 46, Issue D1, 4 January 2018, Pages D413–D418, DOI: 10.1093/nar/gkx1015



