Newsletter DPT Nro. 62
ISSN 2618-236X
Julio / 2021
CUESTIONES DE INTERES
El universo del microbioma
Hallazgos, oportunidades y retrospecciones
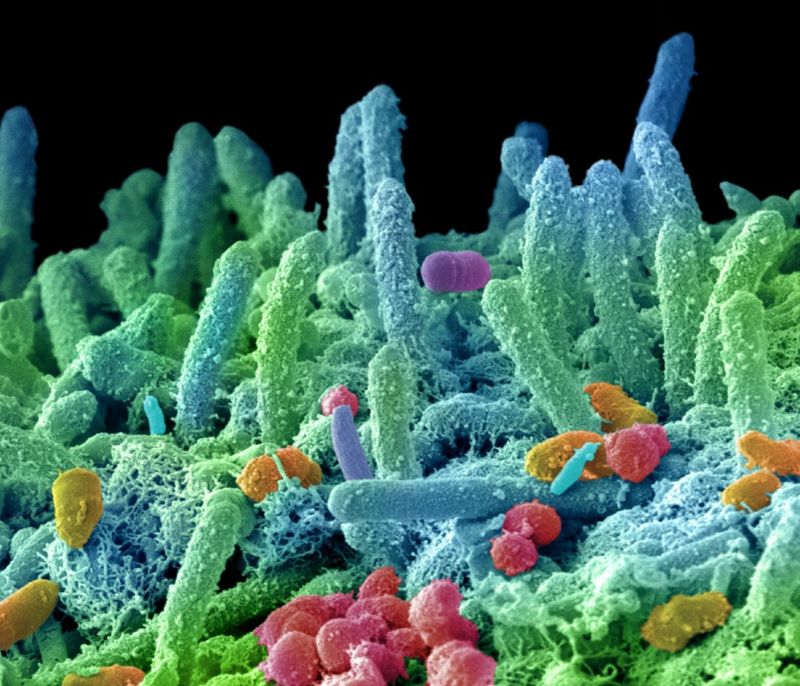
El término microbioma abarca tanto la microbiota (comunidad de microorganismos: bacterias, arqueas, protozoos, hongos y algas) como su entorno de actividad (estructuras microbianas, elementos genéticos móviles, genomas y distintos metabolitos), las condiciones ambientales y su interrelación con el organismo hospedador.
Se trata de una relación netamente simbiótica entre huésped y hospedador. El microbioma, en las proporciones adecuadas, ayuda a mantener un cierto bioequilibrio en nuestros cuerpos, así como en cada elemento existente en el suelo, el aire, el polvo o las aguas.
Cada individuo humano alberga unos 100 billones de bacterias de unas 400 especies distintas. En el aparato digestivo, por ejemplo, la concentración de microorganismos por mililitro de contenido va creciendo a lo largo del intestino delgado (con una media de 105 bacterias/ml), mientras que es mucho mayor en el colon, donde se alcanzan concentraciones de hasta 1.012 bacterias/ml). La población viva del colon supone más del 95% de la microbiota del hospedador humano; puede alcanzar -en conjunto- un peso variable de 300 a 600 gramos.
Si bien el término “microbioma” apareció en la literatura científica en 1988, solo recientemente se fue plasmando una comprensión integradora de qué son los microbiomas, cómo funcionan, su importancia y su impacto en la salud y la enfermedad. Las modernas tecnologías de secuenciación de ADN vienen aportando una mirada más detallada -a nivel genético- del “mundo microbiano”, ayudando a comprender qué organismos están presentes, sus posibles funciones y su equilibrio funcional.
Ciertas dietas y el uso excesivo de antimicrobianos pueden alterar el delicado equilibrio del micribioma humano. Por su parte, la agricultura intensiva y el uso de productos químicos pueden afectar negativamente a los microbiomas del suelo y, en consecuencia, también a la salud de plantas y animales. Algo similar ocurre con el aire y las aguas.
La presente reseña comprende cuatro (4) artículos. En el primero (1) se analiza el papel del microbioma en la salud y la enfermedad, y se destaca su potencial como herramienta de diagnóstico y estrategia terapéutica. En el segundo (2.1) (2.2) y (2.3.) se revelan hallagos sobre el microbioma de nuestros ancestros milenarios. El tercero (3.1.) (3.2.) se refiere a un estudio global de microbioma urbano que identifica miles de nuevas especies. El cuarto (4.1) y (4.2.) señala interesantes hallazgos sobre el microbioma en comunidades remotas de Sudamérica.
1.- Primer artículo: Relevancia del microbioma como herramienta de diagnóstico y estrategia terapéutica (1)
Si bien las interacciones microbioma-hospedador juegan un papel clave en varios procesos biológicos (como nutrición y metabolismo), su intrincada naturaleza conlleva también riesgos en términos de enfermedad. Por ejemplo, durante la última década ha habido un creciente interés en la relación directa entre microbioma y cáncer, así como en cómo los microbios pueden afectar la respuesta a la inmunoterapia del cáncer.
Asimismo, el microbioma fue objeto de un intenso escrutinio, por parte de neurocientíficos, frente a una explosión de datos que conectan: (a) las bacterias intestinales con enfermedades del cerebro, y (b) el cerebro con las células nerviosas del intestino. Los avances en la capacidad para secuenciar el microbioma permitieron vincular las firmas del microbioma con diferentes enfermedades cerebrales, tales como las de Alzheimer y Parkinson, el accidente cerebrovascular isquémico y los trastornos del espectro autista. Las investigaciones futuras sobre el eje cerebro-intestino-microbioma deberán desentrañar con más detalle los vínculos causales propuestos.
Un número creciente de estudios identifican ciertos niveles de microbios como asociados a enfermedades o condiciones particulares. Estas firmas microbianas podrían ayudar a diagnosticar enfermedades -más precozmente y con mayor precisión- con base en microbiomas provenientes de diversas mucosas (respiratorias, digestivas, génito-urinarias, etc.). La piel (el órgano más grande y la primera barrera del cuerpo humano) también alberga una amplia variedad de microbios que pueden proporcionar información de diagnóstico y pronóstico de diversas afecciones cutáneas.
Respecto de las estrategias terapéuticas, el microbioma puede ayudar al hospedador a combatir la enfermedad. Por ejemplo, recientemente se descubrió que ciertas bacterias específicas presentes en la microbiota intestinal pueden ayudar al sistema inmunológico a generar una respuesta antitumoral. Un estudio reciente demostró que al combinar la inmunoterapia con una terapia microbiana específica es posible estimular la respuesta inmune contra ciertos cánceres (melanoma, vejiga y colorrectal).
Además de aprovechar el microbioma en terapéutica, se están examinando los microbiomas de diversos organismos en un intento por identificar compuestos bioactivos con propiedades anticancerígenas. Existe también un creciente interés en el desarrollo de terapias vivas, mediante la alteración de bacterias para producir compuestos terapéuticos.
2.- Segundo artículo: Hallazgos emergentes del análisis del microbioma de nuestros ancestros milenarios (2.1.) (2.2.) (2.3.)
Dado que las bacterias que intervienen al digerir los alimentos que ingerimos se incorporan a las excretas, el disponer de coprolitos (paleofeces, heces conservadas) de 1.000 años de antigüedad puede aportar información sobre: (a) la composición de los ecosistemas bacterianos intestinales de nuestros ancestros humanos, y (b) cómo fueron alterados dichos ecosistemas por mutaciones provenientes del saneamiento, productos químicos (fertilizantes, insecticidas, herbicidas), alimentos procesados, antibióticos y otros factores asociados a la industrialización.
Los artículos aquí reseñados se refieren al análisis –por parte de un equipo internacional dirigido por el Joslin Diabetes Center afiliado a la Escuela de Medicina de Harvard- de coprolitos humanos hallados en la parte trasera de refugios rocosos en Utah y México. Los resultados del análisis sugieren que, durante el último milenio, el intestino humano experimentó un “evento de extinción”, perdiendo docenas de especies y degradando significativamente su diversidad. ¿Qué factores explican ese cambio? ¿Fueron los alimentos, su procesamiento, los productos químicos, los antibióticos, el saneamiento, las medidas preventivas?
Mediante la rehidratación de coprolitos pudieron recuperarse microbiomas provenientes de intestino humano milenario, que son notablemente más diversos que los actuales, incluyendo docenas de especies hoy desconocidas. Los nuevos datos muestran que: (a) todos los actuales habitantes del planeta han sido afectados por cambios en su microbioma, incluyendo a las poblaciones cazadoras y recolectoras relativamente aisladas de las comunidades civilizadas, y (b) dado que los cambios fueron mayores durante el pasado reciente, es posible que nuestros cuerpos no hayan tenido aún tiempo suficiente para adaptarse.
El estudio de los microbios intestinales que se encuentran en las paleofeces puede ofrecer pistas para combatir la diabetes, así como a enfermedades autoinmunes y alérgicas que hoy afectan a las sociedades industrializadas.
Las diferencias entre las poblaciones de microbiomas fueron sorprendentes. Se verificó que casi el 40% de las especies microbianas antiguas nunca se habían hallado en microbiomas actuales. Por ejemplo, una bacteria conocida como Treponema succinifaciens –presente en la totalidad de los microbiomas antiguos- no estaba en ningún microbioma analizado de las actuales poblaciones. Pero también se determinó que los microbiomas antiguos coinciden, en mayor medida, con los microbiomas de poblaciones modernas “no industrializadas”.
Concluye señalando que, a medida que se avanza hacia la industrialización alimentaria y más hacia una dieta de supermercado, se pierden muchos nutrientes que ayudan a mantener un microbioma más diverso.
3.- Tercer artículo: Un estudio global de microbioma urbano identifica miles de nuevas especies (3.1.) (3.2.) (3.3.) (3.4.) (3.5.)
Según el Consorcio Internacional Metagenomics and Metadesign of Subways and Urban Biomes (MetaSUB), un estudio global para rastrear microbios en muestras de ADN en sistemas de transporte público y hospitales de todo el mundo permitió descubrir que alrededor del 45% de los microbios identificados no coincidían con ninguna especie conocida: 10.928 virus y 1302 bacterias recolectados en las muestras no estaban presentes en ninguna base de datos de referencia.
Para el estudio se recolectaron cerca de 5.000 muestras durante 3 años (entre 2015 y 2017) en 60 ciudades de 32 países y 6 continentes. Los investigadores analizaron las muestras de ADN utilizando una técnica de secuenciación genómica denominada “secuenciación de escopeta” para detectar la presencia de diversos microbios (bacterias, arqueas y virus) que utilizan el ADN como material genético. Cabe señalar que los métodos de detección de ADN utilizados en este estudio prepandémico no habrían detectado otros tipos de virus que usan ARN como material genético, como el SARS-CoV-2, que causa COVID-19.
Con base en la información resultante del muestreo puede inferirse, con un 90% de precisión, dónde vive una persona, simplemente secuenciando el ADN en sus zapatos. Un microbioma contiene ecos moleculares del lugar donde fue recolectado (población, densidad, elevación, clima, proximidad a bosques o a cursos de agua). Los hallazgos sobre estas firmas distintas podrían permitir futuros estudios forenses.
Las muestras microbianas se comenzaron a recolectar y analizar en el sistema de metro de la ciudad de Nueva York en 2013. Una vez publicados los primeros hallazgos (“PathoMap”) el equipo fue contactado por investigadores de todo el mundo para realizar estudios similares en sus propias ciudades.
El interés internacional inspiró la creación de MetaSUB, que hoy se ha expandido para recolectar muestras de ARN y ADN del aire, agua y aguas residuales, además de superficies duras. El grupo también supervisa proyectos como el Día Global de Muestreo de Ciudades (gCSD), que se celebra el 21 de junio de cada año, y realizó estudios de amplio alcance, incluido un análisis microbiano integral de Río de Janeiro antes, durante y después de los Juegos Olímpicos de 2016.
Este catálogo sistemático global de microbiomas urbanos representa un atlas metagenómico para comprender la ecología, la virulencia y la resistencia a los antibióticos de las comunidades microbianas específicas de cada ciudad. Aunque las muestras se recolectan en todo el mundo, gran parte del análisis se realiza en Weill Cornell Medicine de New York City. Para el análisis y ensamblaje de secuencias se utilizan las supercomputadoras Bridges and Bridges-2, Extreme Science and Engineering Discovery Environment (XSEDE) en el Pittsburgh Supercomputing Center. Los investigadores de MetaSUB en Suiza construyeron un portal global de secuencias de ADN con capacidad de búsqueda (MetaGraph) que indiza todas las secuencias genéticas conocidas (incluidos los datos de MetaSUB).
Este campo de investigación tiene importantes implicaciones para detectar brotes de infecciones conocidas y desconocidas, así como para identificar y estudiar la aparición de microbios o de cepas resistentes a los antibióticos en diferentes entornos urbanos. También podría conducir al descubrimiento de nuevos antibióticos, así como de otras moléculas que podrían desarrollarse como fármacos. Los descubrimientos sobre nuevas especies microbianas también podrían contribuir al desarrollo de nuevas herramientas y enfoques de laboratorio, como formas novedosas de utilizar la herramienta de edición molecular conocida como CRISPR.
Se concluye destacando que los sistemas urbanos de transporte masivo son una fuente inexplorada y fenomenal de biodiversidad. Las especies descubiertas tienen potencial para la investigación de medicamentos, y el mapeo y monitoreo de microbiomas urbanos a gran escala ayudaría a detectar precozmente patógenos emergentes.
Cuarto artículo: Hallazgos sobre el microbioma en comunidades remotas de Sudamérica (4.1) y (4.2.)
El artículo se refiere a las experiencias y hallazgos de la Dra. María Gloria Domínguez, de la Rutgers University, en sus investigaciones sobre el microbioma en comunidades indígenas aisladas, cazadoras y recolectoras. Sus preguntas de investigación, respecto de dichos microbiomas, son: ¿Por qué hallamos esto? ¿De dónde provino? ¿Cuándo y cómo se plasmó esta simbiosis?”
Los estudios de la Dra. Domínguez y su equipo sobre algunas poblaciones indígenas reflejan una notable diversidad de microbiota. Por ejemplo, tras analizar el microbioma bacteriano (fecal, oral y cutáneo) de integrantes de una cierta comunidad remota, se halló que “albergan un microbioma con la mayor diversidad de bacterias y funciones genéticas jamás reportada en un grupo humano”. Pero aun con su aislamiento y “sin exposición conocida a antibióticos, albergan bacterias que portan genes funcionales de resistencia a los antibióticos, incluidas las que confieren resistencia a los antibióticos sintéticos”. Destacan que los resultados sugieren que “la occidentalización afecta significativamente a la diversidad del microbioma humano”.
Concluye destacando que el gradiente de urbanización es clarísimo: a medida que las personas viven en ciudades y adoptan el estilo de vida industrializado, acogen “muchísimas prácticas” que son antimicrobianas. Y no sólo se trata de hábitos de higiene, sino del consumo de antibióticos, del uso de sustancias antibacterianas y de conservantes. “En esta cultura de dietas procesadas y conservadas, también estamos comiendo mucho antimicrobiano”.
Referencias:
(1) “Inside the microbiome”. Written by The Editors at Technology Networks. Sponsored by ThermoFisher Scientific. 11/01/2021
Acceso al artículo completo:
(2.1.) Fuente primaria: “Reconstruction of ancient microbial genomes from the human gut” Marsha C. Wibowo, Zhen Yang, Maxime Borry, Alexander Hübner, Kun D. Huang, Braden T. Tierney, Samuel Zimmerman, Francisco Barajas-Olmos, Cecilia Contreras-Cubas, Humberto García-Ortiz, Angélica Martínez-Hernández, Jacob M. Luber, Philipp Kirstahler, Tre Blohm, Francis E. Smiley, Richard Arnold, Sonia A. Ballal, Sünje Johanna Pamp, Julia Russ, Frank Maixner, Omar Rota-Stabelli, Nicola Segata, Karl Reinhard, Lorena Orozco, Christina Warinner, Meradeth Snow, Steven LeBlanc & Aleksandar D. Kostic. Nature (2021) Article. Open Access. Published: 12 May 2021. DOI: 10.1038/s41586-021-03532-0
(2.2.) Fuente secundaria 1: “Did Ancient People Have More Diverse Gut Microorganisms?”. News. May 13, 2021. Genomics Reserach from Technology Networks.| Original story from University of Montana
(2.3.) Fuente secundaria 2: “Piles of ancient poop reveal ‘extinction event’ in human gut bacteria” By Andrew Curry. Science. American Association for the Advancement of Science (AAAS). Archeoilogy, Evolution. May. 12, 2021. DOI: 10.1126 / science.abj4390
(3.1.) Fuente primaria: “A global metagenomic map of urban microbiomes and antimicrobial resistance” David Danko, Daniela Bezdan, Evan E. Afshin, …, Sibo Zhu, Christopher E. Mso, The International MetaSUB Consortium. Cell. Open Access, Published: May 26, 2021. DOI: 10.1016/j.cell.2021.05.002
(3.2.) “Global microbiome study identifies thousands of new species” Technology Networks. 27/05/2021 | Weill Cornell Medicine
(3.3.) “Cities have their own distinct microbial fingerprints” By Cathleen O’Grady. Science – American Association for the Advancement of Science (AAAS)- 26 May 2021
(3.4.) Mapeo METASUB: Metagenómica y metadiseño del metro y los biomas urbanos
(3.5.) Datos Abiertos de ADN– “MetaGraph: Ultra Scalable Framework for DNA Search, Alignment, Assembly”
(4.1.) Fuente primaria: “A Microbial Anthropologist in the Jungle” By Maria Gloria Dominguez-Bello. Cell. Leading Edge. Stories. 2016
(4.2.) Fuente secundaria: “Los fascinantes hallazgos sobre el microbioma humano de una bióloga venezolana que estudia comunidades remotas de Sudamérica” Por Margarita Rodríguez, BBC News Mundo. 18 mayo 2021


