Newsletter DPT Nro. 76
ISSN 2618-236X
Septiembre / 2022
NOTICIAS CIENTIFICAS
NOTICIAS CIENTIFICAS INTERNACIONALES
Es posible inferir la estructura de casi todas las proteínas conocidas por la ciencia
Con herramientas de Inteligencia Artificial
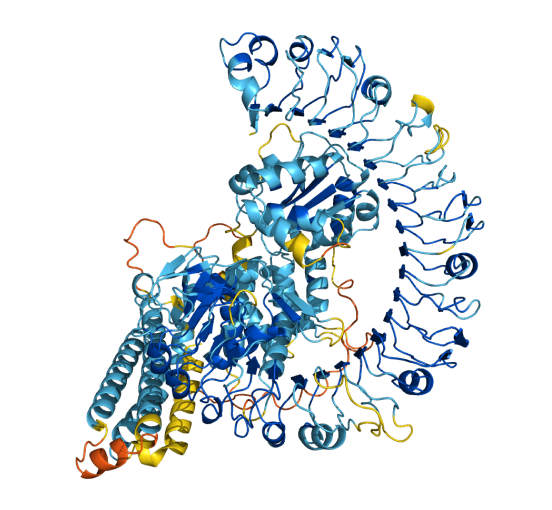
Mediante un modelo de inteligencia artificial (IA), la empresa británica DeepMind (propiedad de Google) y el Instituto Europeo de Bioinformática del Laboratorio Europeo de Biología Molecular (EMBL-EBI) lograron inferir las estructuras tridimensionales de casi todas las proteínas conocidas y catalogadas, para ofrecerlas -de forma gratuita y abierta- en la base de datos de estructuras de proteínas AlphaFold.
Demis Hassabis, fundador y director ejecutivo de DeepMind, señala: “Nos ha sorprendido la velocidad con la que este desarrollo se convirtió en una herramienta esencial para cientos de miles de investigadores en laboratorios y universidades de todo el mundo”. “Desde la lucha contra enfermedades hasta la contaminación por plásticos, AlphaFold ya permitió significativos impactos en algunos desafíos globales”… “Nuestra expectativa es que esta base de datos ampliada ayude a muchos más investigadores en su trabajo y abra vías de descubrimiento completamente nuevas”.
DeepMind y EMBL-EBI lanzaron AlphaFold en julio de 2021, con más de 350.000 predicciones de estructuras de proteínas, incluido el proteoma humano completo. Las actualizaciones posteriores permitieron adicionar otros relevantes repositorios. En poco más de un año, más de 1.000 artículos científicos citaron a esta base de datos y más de 500.000 investigadores de más de 190 países accedieron a AlphaFold para visualizar más de 2 millones de estructuras tridimensionales de proteínas. La comunidad científica también aprovechó este sistema para crear y adaptar herramientas como Foldseek y Dali, que permiten buscar entradas de proteínas concretas. También se adoptaron ideas centrales de aprendizaje automático, que sustentan este programa, para desarrollar nuevos algoritmos o aplicarlos en áreas como la predicción de la estructura del ARN o la creación de nuevos modelos proteicos. Según sus promotores, AlphaFold contribuyó también a mejorar las capacidades para combatir la contaminación por plásticos, obtener información sobre la enfermedad de Parkinson, mejorar la salud de las abejas melíferas, comprender cómo se forma el hielo, abordar patologías desatendidas como la enfermedad de Chagas y la leishmaniasis, así como a explorar -con mayor precisión- la evolución humana.
DeepMind y EMBL-EBI continuarán actualizando el AlphaFold para mejorar sus características y funcionalidad en respuesta a los comentarios de los usuarios. El acceso a la base de datos continuará siendo abierto, bajo una licencia Creative Commons, y las descargas masivas estarán disponibles a través de Google Cloud.
Fuente primaria: “AlphaFold: Protein Structure Database: Developed by DeepMind and EMBL-EBI”. 2022
Fuente secundaria 1: “AlphaFold: Protein Structure Database” Developed by DeepMind and EMBL-EBI. European Bioinformatics Institute (EMBL-EBI)
Fuente secundaria 2: “La IA predice la estructura de casi todas las proteínas conocidas por la ciencia”. Boletín SINC. 29/7/2022


